
beyond reason
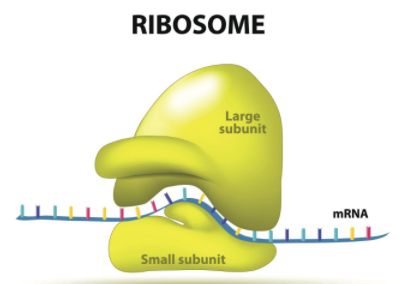
리보솜(영어: ribosome)은 아미노산을 연결하여 단백질 합성을 담당하는 세포소기관으로 리보솜 RNA와 단백질로 이루어져 있다. 리보솜은 대단위체와 소단위체로 분리되어 있으며, 두 단위체가 결합하여 단백질 합성을 수행한다.[1]
리보솜은 지름이 약 20 nm(200 Å)이고 65%의 리보솜 RNA와 35%의 리보솜 단백질로 구성되어 있다. 고균, 진정세균 및 진핵생물의 리보솜은 서로 다른 다양한 단백질과 RNA로 구성되어 있다. 리보솜은 전령 RNA의 코돈을 번역하여 운반 RNA에 연결된 아미노산을 배열하여 단백질을 형성한다. 리보솜은 별도의 막이 없는 세포소기관이다.
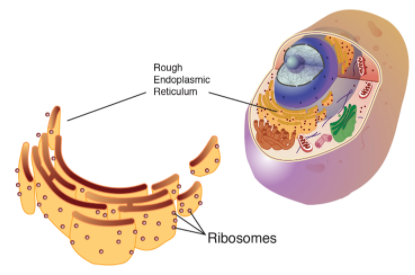
단백질과 RNA로 이루어진 리보솜과 조금다른 리보자임은 효소처럼 작용하는 RNA분자이다. 이것에 의해 RNA 세계 기원설이 제기되기도 한다. 단백질을 형성하지 않는 동안 RNA는 내부 원형질인(세포기질cytosol) 때에는 RNA로부터 구성되는데, 이는 RNA 세계의 흔적으로 추정된다.[2] 리보솜은 1950년대 중반 루마니아 생물학자인 게오르그 에밀 팔라데에 의해 최초로 관찰되었다. 팔라데는 전자현미경을 이용하여 세포를 관찰하던 중 리보솜을 발견하였고, 이 공로로 1974년 노벨 생리학·의학상을 수상하였다.[3] 리보솜이라는 이름은 1958년 리처드 B. 로버트가 명명하였다.
| “ | 지금까지 학계에서 "마이크로솜"이라 불리고 있는 단백질 형성 세포소기관을 "리보솜"이라 부르자고 제안합니다. 이 소체는 리보핵산단백질과 RNA로 이루어져 있기 때문입니다. 리보솜 총량을 100으로 할 때 리보핵산단백질은 35를 차지하고 있습니다. | ” |
— 리처드 B. 로버트, 〈세포소기관과 단백질 효소〉[4] |
20세기 중반에 리보솜의 분자 구조와 기능이 발견된 이후 현재까지 활발한 연구가 계속되고 있다. 실제로, 아다 요나스(이스라엘), 벤카트라만 라마크리시난(미국)과 토머스 스타이츠(미국)는 리보솜의 기능과 구조에 관한 연구로 2009년 노벨 화학상을 수상하기도 하였다.
생성
세균의 리보솜은 세포질에 있는 유전자인 오페론을 전사하여 생성된다. 진핵생물의 경우 세포핵 안에 있는 핵소체에서 4 종류의 리보솜 RNA가 만들어지며 리보솜 단백질과 결합되어 리보솜이 생성된다.
단백질 생성과정
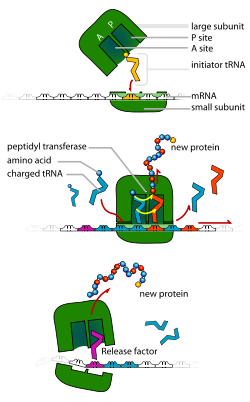
리보솜에서 이루어지는 단백질 형성과정
리보솜이 mRNA의 코돈을 인식하여 단백질을 합성하는 과정을 번역이라 한다. 리보솜에서 이루어지는 단백질 생성과정은 다음과 같다.[5][6]
- 준비
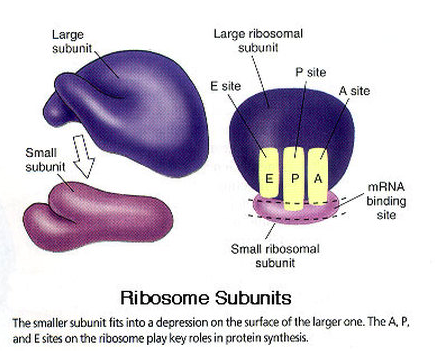
- 리보솜의 소단위체와 대단위체가 결합한다. 리보솜에는 운반 RNA가 들어설 수 있는 부위가 두 곳이 있는데 운반 RNA가 들어오는 쪽을 A 자리, 나가는 쪽을 P 자리라 한다.
- 연장 과정
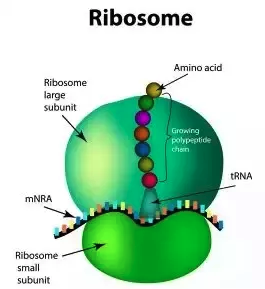
- 코돈 인식 : 폴리펩타이드를 달고 있는 P 자리의 tRNA가 코돈에 붙어있다. A 자리에 있는 전령 RNA의 코돈과 상보성을 갖는 안티코돈을 가진 운반 RNA가 아미노산을 가져와 결합한다.
- 펩타이드 결합 형성 : P 자리에 있는 기존의 폴리펩타이드와 A 자리에 있는 새로운 운반 RNA의 아미노산 사이에 펩타이드 결합이 형성된다. 이때 아미노아실 결합이 P 자리의 운반 RNA에서 새로운 폴리펩타이드 쪽으로 옮겨간다.
- 변환 : P 자리에서 자유로워진 운반 RNA가 빠져나간다. 이제 전령 RNA가 한칸 움직일 수 있게 되고 코돈이 바뀐다.
- 반복 : A 자리의 폴리펩타이드-운반 RNA 중합분자가 P 자리로 옮겨가면서 A 자리가 비워진다. 비워진 A 자리에는 바뀐 코돈과 상보적인 운반 RNA가 새롭게 결합된다. 위 과정이 계속 반복되면서 폴리펩타이드 사슬이 길어진다.
- 종료
- 종료 코돈에 종결 운반 RNA가 결합되면 합성되던 폴리펩타이드가 떨어져 나가면서 단백질 합성이 끝난다.
리보솜에서 일어나는 번역 과정의 에너지원 는 GTP의 가수분해이다.
 |
| 단백질 형성의 진행 |
start codon
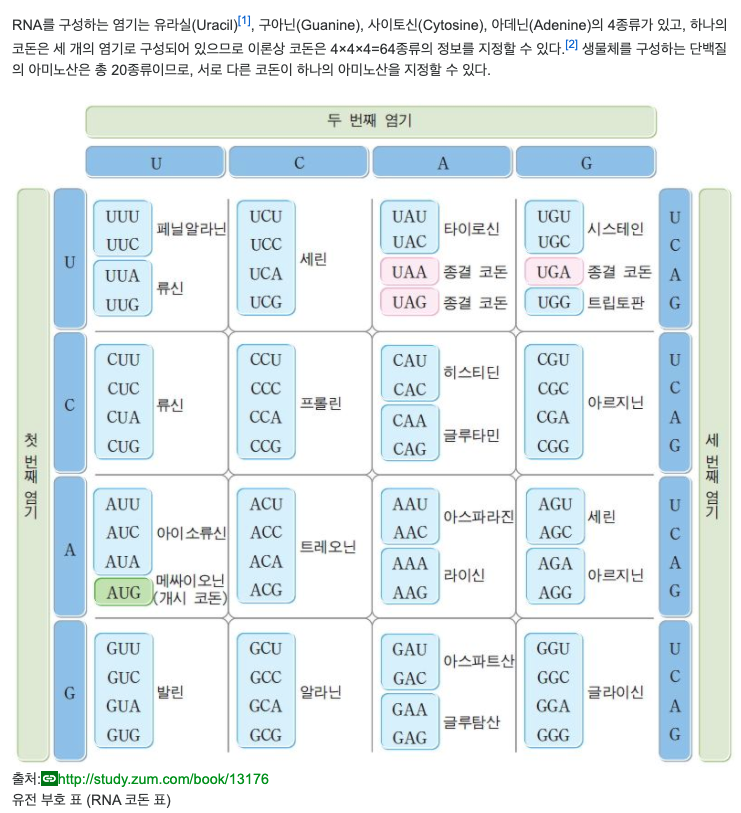
'시작 코돈'이라고도 하며, 5'-AUG-3'이다. 하지만 예외적으로 일부 박테리아에서 NUG(GUG,TUG,CUG)의 변형된 개시 코돈이 사용된다. 해당 코돈은 진핵 생물에서는 아미노산 메싸이오닌(Methionine, Met)을, 원핵생물에서는 변형된 메티오닌인 N-포르밀메싸이오닌(N-Formylmethionine, fMet)을 지정한다. 이와 함께 이 코돈은 mRNA가 리보솜과 결합해 단백질 번역을 시작하도록 하는 역할 또한 수행하며, 이 때문에 개시 코돈이라는 이름으로도 불린다. 유전자에서 염기 한두 개가 변하는 SNP(Single Nucleotide Polymorphism)이 개시 코돈에서 발생 시 만들어져야 할 단백질이 아예 생성되지 않게 되므로 크나큰 영향을 미치게 된다.
Stop Codon, Nonsense Codon
단백질 번역의 끝을 알리는 코돈. UAA, UAG, UGA의 세 종류가 있다. 우아아 우악 우가[4]
종결 코돈에는 대응하는 tRNA가 없고 대신 '종결 인자'라는 단백질이 붙으며[5], 번역 과정에서 종결 코돈에 도달하면 리보솜의 두 단위체가 분리되어 번역이 종결된다. 아미노산을 암호화하지 않는 코돈이라는 의미로 nonsense codon이라고도 불린다. 아미노산을 암호화하는 나머지 61가지 코돈은 반대로 sense codon. 멀쩡한 코돈이 돌연변이를 일으켜 종결 코돈이 되어 단백질 합성을 종결해 버리는 경우를 nonsense mutation이라고 부른다. 세 종결 코돈은 별칭이 있는데, UAA는 ochre codon, UAG는 amber codon, UGA는 opal codon이라고도 불린다. 이에 따라 앞서 말한 nonsense mutation에서 코돈이 UAA로 변이한 경우를 ochre mutation, UAG로 변이하면 amber mutation, UGA는 opal mutation이라고 부른다. 특히 UAG 종결 코돈은 비천연 아미노산을 이용한 단백질 합성에서 단골로 쓰인다.
The start codon is the first codon of a messenger RNA (mRNA) transcript translated by a ribosome. The start codon always codes for methionine in eukaryotes and Archaea and a modified Met (fMet) in bacteria, mitochondria and plastids. The most common start codon is AUG (i.e., ATG in the corresponding DNA sequence). The start codon is often preceded by a 5' untranslated region (5' UTR). In prokaryotes this includes the ribosome binding site.
Alternative start codons[edit]
Alternative start codons are different from the standard AUG codon and are found in both prokaryotes (bacteria and archaea) and eukaryotes. Alternate start codons are still translated as Met when they are at the start of a protein (even if the codon encodes a different amino acid otherwise). This is because a separate transfer RNA (tRNA) is used for initiation.[1]
Eukaryotes[edit]
Alternate start codons (non-AUG) are very rare in eukaryotic genomes. However, naturally occurring non-AUG start codons have been reported for some cellular mRNAs.[2] Seven out of the nine possible single-nucleotide substitutions at the AUG start codon of dihydrofolate reductase were functional as translation start sites in mammalian cells.[3] In addition to the canonical Met-tRNA Met and AUG codon pathway, mammalian cells can initiate translation with leucine using a specific leucyl-tRNA that decodes the codon CUG.[4][5]
Candida albicans uses a CAG start codon.[6]
Prokaryotes[edit]
Prokaryotes use alternate start codons significantly, mainly GUG and UUG.[7]
E. coli uses 83% AUG (3542/4284), 14% (612) GUG, 3% (103) UUG[8] and one or two others (e.g., an AUU and possibly a CUG).[9][10]
Well-known coding regions that do not have AUG initiation codons are those of lacI (GUG)[11][12] and lacA (UUG)[13] in the E. coli lac operon. Two more recent studies have independently shown that 17 or more non-AUG start codons may initiate translation in E. coli.[14][15]
Mitochondria[edit]
Mitochondrial genomes use alternate start codons more significantly (AUA and AUU in humans).[7] Many such examples, with codons, systematic range, and citations, are given in the NCBI list of translation tables.[16]
Standard genetic code
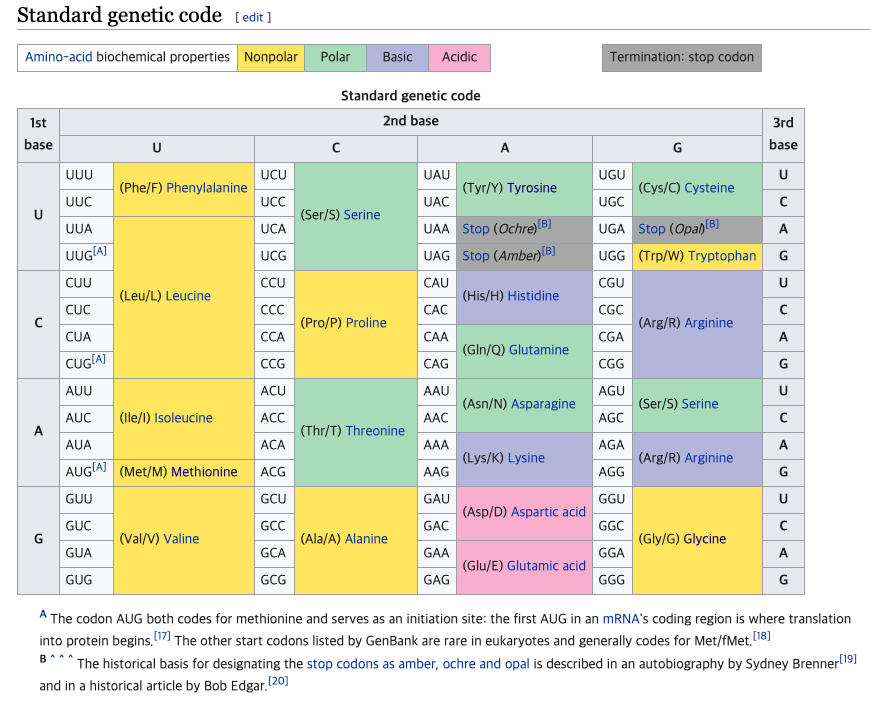
A The codon AUG both codes for methionine and serves as an initiation site: the first AUG in an mRNA's coding region is where translation into protein begins.[17] The other start codons listed by GenBank are rare in eukaryotes and generally codes for Met/fMet.[18]B ^ ^ ^ The historical basis for designating the stop codons as amber, ochre and opal is described in an autobiography by Sydney Brenner[19] and in a historical article by Bob Edgar.[20]
Engineered start codons
Engineered initiator tRNAs (tRNAfMet2 with CUA anticodon) have been used to initiate translation at the amber stop codon UAG.[21] This type of engineered tRNA is called a nonsense suppressor tRNA because it suppresses the translation stop signal that normally occurs at UAG codons. One study has shown that the amber initiator tRNA does not initiate translation to any measurable degree from genomically-encoded UAG codons, only plasmid-borne reporters with strong upstream Shine-Dalgarno sites [22].